本网讯(人工智能学院)近日,安徽大学人工智能学院孙长银教授、叶盛教授团队与中国科学技术大学江俊教授团队合作,成功开发出一套全新的人工智能计算框架,能够从二维红外光谱中直接重建与阿尔茨海默症、帕金森症、Ⅱ型糖尿病及甲状腺素淀粉样变性等重大疾病密切相关的蛋白质动态三维结构。该研究成果以“A Machine Learning Protocol for Predicting Structural Distributions of Amyloid-Forming Proteins from 2D IR Spectra” 为题,发表在国际知名学术期刊《美国国家科学院院刊》(PNAS,www.pnas.org/doi/10.1073/pnas.2522772122)上。
蛋白质错误折叠被认为是多种神经退行性疾病的核心致病机制。然而,这些与疾病密切相关的易聚集片段具有高度动态性与瞬态特征,传统结构解析手段难以在原子水平上精确捕捉。二维红外光谱能够提供皮秒至飞秒时间尺度下的振动“指纹”,对蛋白质构象变化极为敏感,但如何从复杂的光谱信号中反演出精确的三维结构,一直是困扰该领域的重大科学难题。
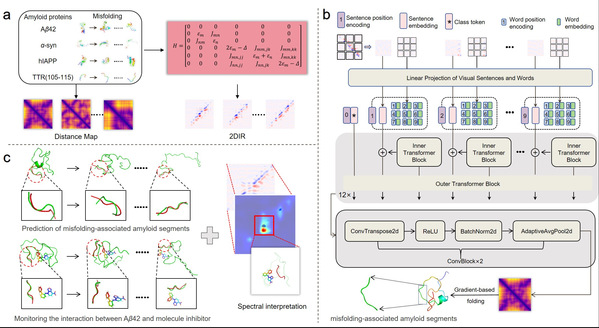
图:人工智能揭示淀粉样蛋白错误折叠路径中的结构演化规律
针对这一挑战,研究团队创新性地提出了“光谱驱动的结构建模”理念,融合量子化学计算、深度学习与结构建模技术,构建了大规模蛋白质构象–光谱映射数据库,并训练出可从二维红外光谱预测蛋白质三维结构的机器学习模型。研究结果表明,该方法能够准确重构包括阿尔茨海默症β淀粉样蛋白、α-突触核蛋白、人胰岛淀粉样多肽及运甲状腺素蛋白等多种致病蛋白的关键聚集片段结构。值得特别指出的是,该模型还能够识别小分子抑制剂与蛋白结合引发的构象动态变化,清晰揭示配体结合诱导的结构重组过程。该研究实现了从二维光谱直接解析蛋白原子级结构,为淀粉样蛋白错误折叠的机制研究与抑制剂设计提供了关键方法突破。




